柳叶刀子刊《Lancet Digital Health》报道了北航医学科学与工程学院在肺癌人工智能研究的最新进展:《Mining Whole-lung Information by Artificial Intelligence for Predicting EGFR Genotype and Targeted Therapy Response in Lung Cancer: A Multicohort Study》,该研究提出了全肺分析人工智能模型,可利用CT影像无创预测肺癌患者的基突变状态和接受靶向治疗后的个体化疗效,最终可辅助肺癌患者的靶向治疗决策。该研究使用了1.8万例肺癌患者数据对模型进行训练和验证,利用人工智能技术进一步探索了宏观CT影像与微观基因信息的关联。
该工作属于医工交叉的深入研究,论文第一作者为北航医学科学与工程学院王硕副教授;华西医院余何、甘芸翠博士作为共同一作;共同通讯作者为华西医院呼吸与危重症医学科李为民教授、上海市肺科医院放射科史景云教授、广东省人民医院放射科刘再毅教授、世纪坛医院呼吸与危重症医学科薛新颖教授、大连医科大学附属第二医院呼吸内科王琪教授、云南省肿瘤医院放射科李振辉博士、301医院呼吸与危重症医学科谢菲教授以及北航医学科学与工程学院田捷教授。


肺癌作为死亡率最高的癌症,其中超过一半的患者在确诊时已是晚期,此时,表皮生长因子受体(EGFR)靶向治疗是推荐的一线治疗方案。在靶向治疗过程中,首先需对患者进行EGFR基因突变检测,进而筛选出可以接受靶向治疗的患者。
现有的临床方法通过穿刺取出肿瘤组织再进行基因检测来确定基因突变状态,但穿刺只能获取肿瘤局部组织的信息,由于肿瘤的高度异质性可能产生假阴性结果[1, 2]。此外,一部分晚期肺癌患者其肿瘤所处的位置特殊,或由于癌症转移等因素,难以穿刺到合适的肿瘤组织,导致这部分患者错失靶向治疗的机会。因此,亟需一种无创、方便的EGFR基因突变检测方法,作为对穿刺的补充和辅助。
CT影像作为肺癌诊疗过程中必不可少的工具,提供了很好的无创分析肺癌的手段,且不会受到肿瘤异质性的影响;但仅凭人眼阅片难以对其中蕴含的高维和微观信息进行有效挖掘。人工智能技术的发展则充分利用了大数据的优势,可从影像中挖掘到人眼难以感知的高维微观信息,构建了宏观影像与微观基因信息之间的桥梁[3]。
本文研发了一种全自动的人工智能系统(Fully Automated Artificial Intelligence System, FAIS),在大数据驱动下直接对CT影像进行全自动的分析。FAIS使用了基于肺区引导注意力机制的全肺分析深度学习模型,无需人工辅助和标注图像,对全肺的异常征象进行自动分析,并使用EGFR基因型和基因通路信息对模型进行引导训练;最终,可利用CT影像无创地预测出患者是否存在EGFR基因突变,并且可对患者接受EGFR靶向治疗后的无进展生存期进行预测,进而筛选出靶向治疗的获益人群。
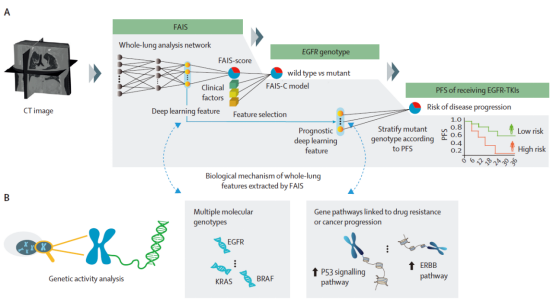
图1. FAIS人工智能系统的流程图
该研究纳入了国内外9个数据集1.8万余例肺癌患者进行大数据的训练和验证。研究发现,肺癌EGFR基因突变会导致肿瘤以外区域的形态学改变,因此,全肺分析比基于肿瘤的分析方法能挖掘到更多的有用信息。在美国TCIA公开数据集的对比实验表明,FAIS所使用的全肺分析方法比传统的基于肿瘤的分析方法性能提升了11.8%。此外,全肺分析方法无需人工勾画肿瘤病灶,更具有可推广性。在TCIA数据集上进行的基因通路分析表明,FAIS从CT影像中提取的全肺特征与ERBB、P53、细胞黏附分子通路、ECM受体相互作用通路等靶向治疗耐药和癌症转移相关的基因通路都存在关联,通过大数据学习进一步揭示了宏观影像与微观基因信息的关系。最终,FAIS在6个测试数据集8000余例肺癌患者中进行EGFR基因突变预测时达到了AUC=0.813的精度;并且能预测患者接受靶向治疗后的无进展生存期,对患者进行预后风险分层。借助FAIS的辅助,可以对原本难以进行穿刺的患者无创地检测出EGFR基因型,并预测其靶向治疗疗效;可有效筛选出靶向治疗获益人群,进而提升肺癌患者的生存率。

图2. FAIS发现EGFR基因突变对宏观CT征象的影响不仅体现在肿瘤内部(A、B),也可能体现在肿瘤周围(C-H)
该研究于2022年3月24日在线发表在柳叶刀子刊Lancet Digital Health上(SCI IF: 24.519),DOI: 10.1016/S2589-7500(22)00024-3。该研究得到国家自然科学基金、科技部重点研发计划等科研基金的资助。
参考文献:
[1]. Gerlinger M, Rowan A J, Horswell S, et al. Intratumor heterogeneity and branched evolution revealed by multiregion sequencing[J]. New England Journal of Medicine, 2012, 366: 883-892.
[2]. Topalian S L, Taube J M, Anders R A, et al. Mechanism-driven biomarkers to guide immune checkpoint blockade in cancer therapy[J]. Nature Reviews Cancer, 2016, 16(5): 275-287.
[3]. Bera K, Braman N, Gupta A, et al. Predicting cancer outcomes with radiomics and artificial intelligence in radiology[J]. Nature Reviews Clinical Oncology, 2021: 1-15.
【Lancet Digital Health原文链接:https://www.thelancet.com/action/showPdf?pii=S2589-7500%2822%2900024-3】